Technology Trend
Archer VARIANTPlex™ 와 FUSIONPlex™ Assays
급성 골수성 백혈병 관련 유전자의
0.1% 미만 빈도의 변이 분석
Focus on4
Gene 心
소개
급성 골수성 백혈병(AML) 에서 미세 잔존 질환의 존재는 암 진행을 이해하는데 중요합니다. 연구에 따르면0.01% allele frequency(AF)의 변이는 급성 골수성 백혈병을 계층화하는데 유용한 것으로 알려져 있습니다[1]. 이와 관련하여 신뢰성 있는 검사를 위해서는 높은 민감도와 높은 특이성이 모두 필요합니다. 따라서 본질적으로 변이검출시 노이즈를 최소화하고 정교하게 변이검출오류를 방지할 수 있는 방법을 선택해야 합니다.
여기에서는 IDT의 Archer VARIANTPlex와 FUSIONPlex Assay(RUO), 그리고 함께 제공되는 소프트웨어인 Archer Analysis를 사용하여 높은 수준의 민감도와 특이성으로 낮은 AF의 변이와 적은 transcript를 사용하여 fusion variant를 검출하는 방법을 보여줍니다. 현재 사용가능한 Archer Assay 및 소프트웨어 솔루션은 AML 관련 변이를 대상으로 하며 여러 NGS platform과 호환됩니다. Archer 기술을 사용하여 주요 AML 관련 0.1% AF 미만의 돌연변이를 검출하였고, Myeloid 관련 fusion variant 를 적은 transcript 수로 검출 하였습니다. 또한, 표적 유전자영역 중 95% 이상이 VARIANTPlex 라이브러리에서 0.1% 미만의 AF에서 변이를 감지하도록 구동되었습니다. 마지막으로, error correction 및 noise filtering 기술을 통해 많은 위양성 변이를 제거하여 위양성 변이 검출율을 줄였습니다.
방법
시료준비를 위해 Myeloid 관련 변이들을 포함하는 상업적으로 이용 가능한 세포주를 사용하였습니다. 해당 변이들을 보유한 1,000ng의 gDNA 또는 200ng의 RNA를 1:100 비율로 정상 gDNA에 희석하였고, 1:2 에서 1:20의 비율로 RNA를 희석하여 분석 감도를 평가 했습니다. 라이브러리는Archer VARIANTPlex AML Focus 또는 FUSIONPlex Pan Heme Assay를 사용하여 제작했습니다. 프로토타입의 VARIANTPlex AML MRD 패널은 50-1,000ng의 NA12877을 사용하여 평가했습니다. VARIANTPlex 라이브러리는 Illumina 및 Element Biosciences sequencer에서 10-50M reads를 사용하여 염기서열분석을 진행했습니다. FUSIONPlex 라이브러리는 Illumina NextSeq 500s에서 4.5~10M reads를 사용하여 염기서열분석을 진행했습니다. Archer Analysis를 사용하여 데이터를 분석했으며, 이 파이프라인에는 read cleaning, deduplication, error correction, variant calling, variant filtering, 그리고 reporting step이 포함되어 있습니다.
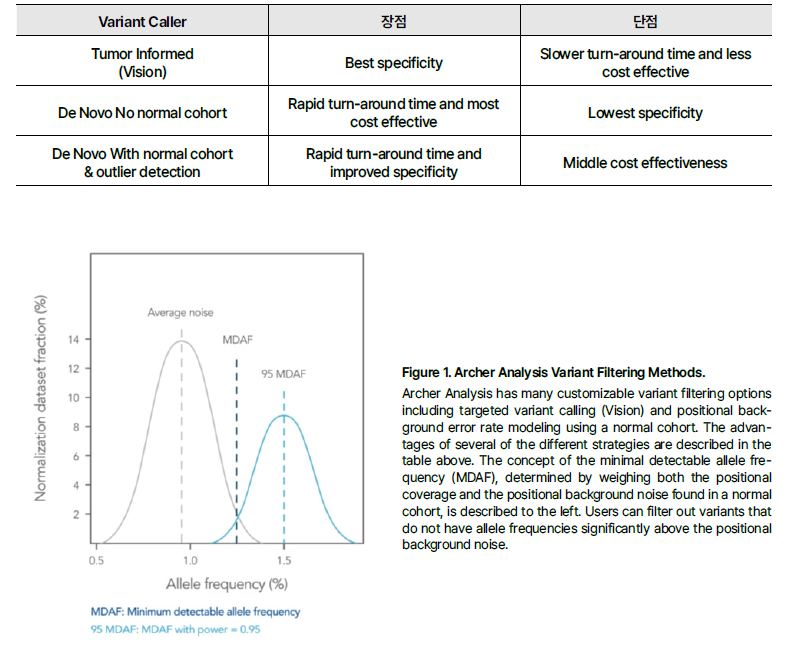
변이 검출 전략
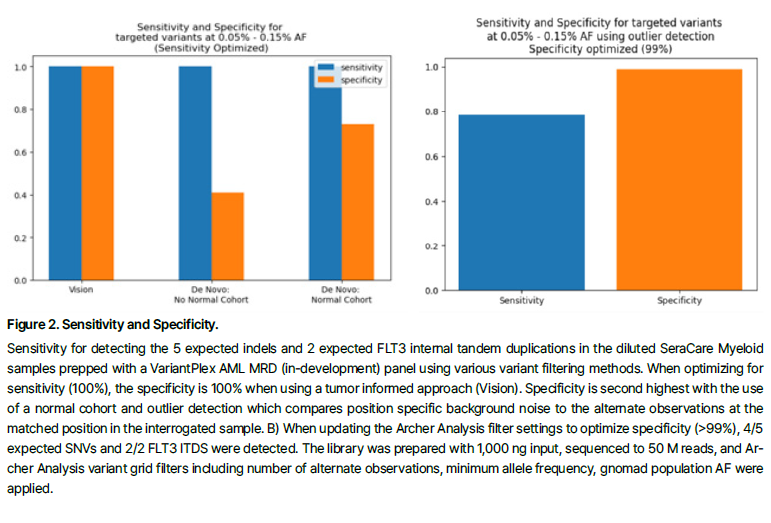
결과 1 : VARIANTPlex Analytical Sensitivity, Coverage, and Error Rates
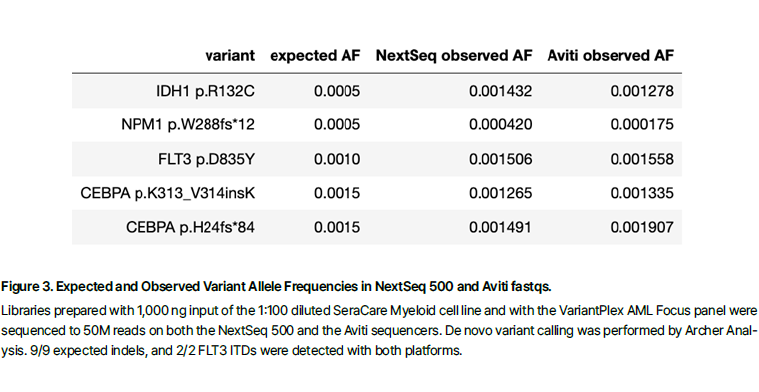
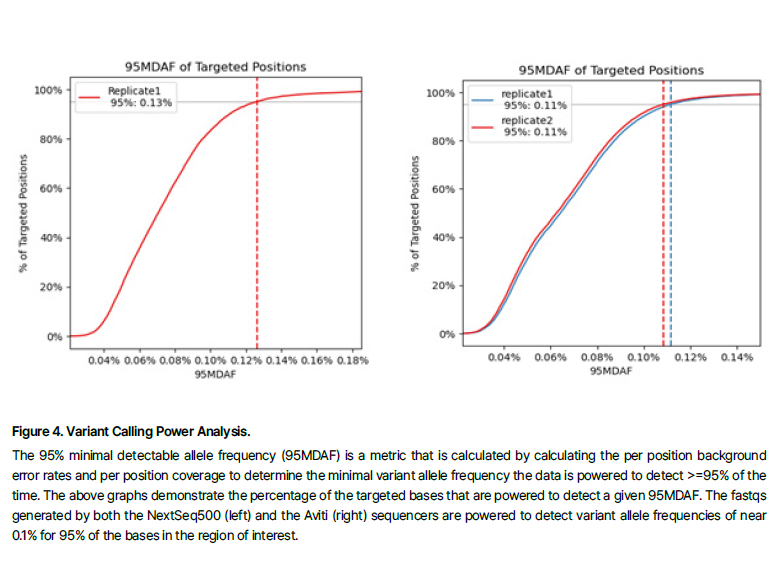
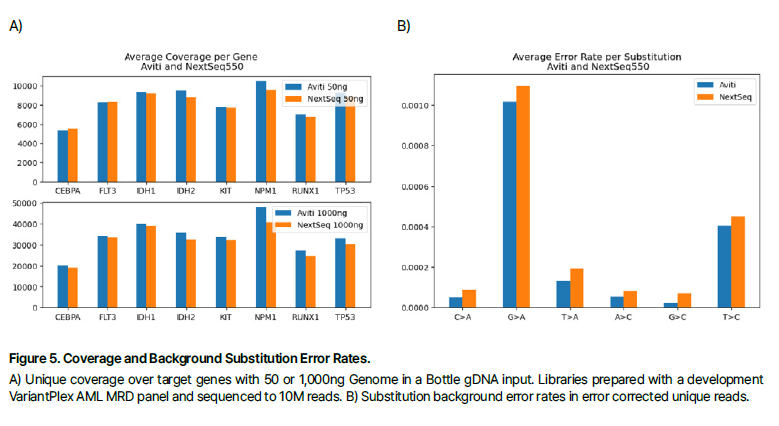
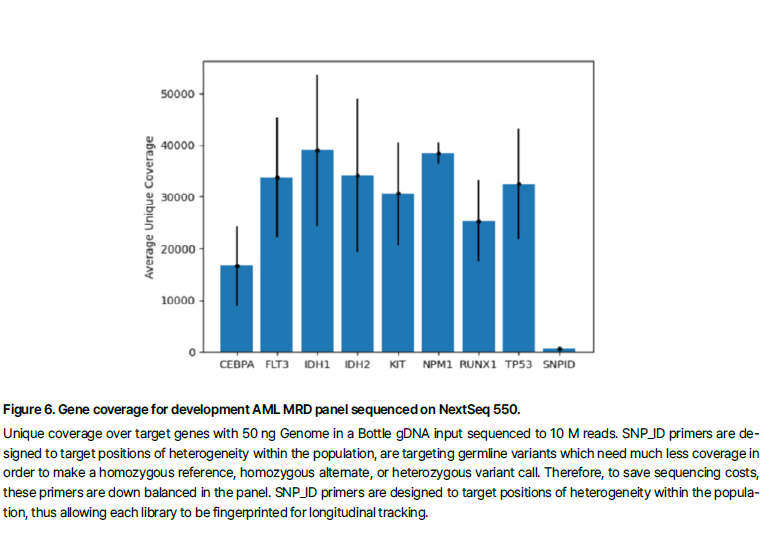
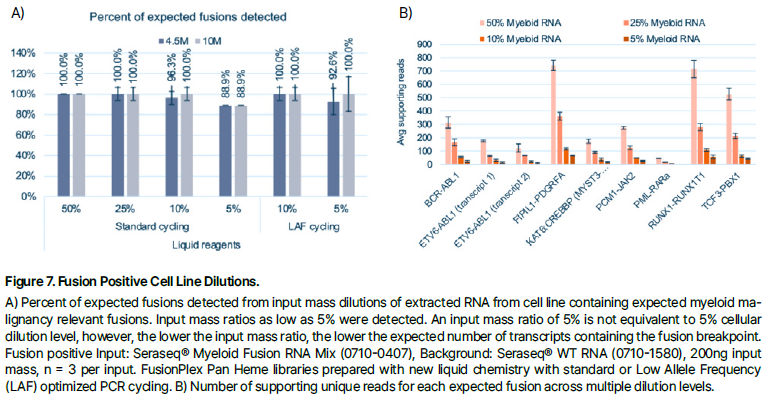
결과 2 : FUSIONPlex Analytical Sensitivity
결론
결론적으로, Illumina 및 Element sequencer 에서 VARIANTPlex AML Focus Assay를 사용하여 0.05% AF의 변이를 검출하는 성능을 입증하였고, FUSIONPlex Pan Heme Assay로 AML 미세잔존질환에서 나타날 수 있는 낮은 AF의 fusion variant를 검출하는 성능을 입증하였습니다.
[References]
[1] Dillon et. al. JAMA. 2023;329(9):745-755.

