한국생명공학연구원
NGS 데이터의 정보분석에서 많은 연구자들이 가장 어려워하는 부분은 (1) Linux 환경에서 구동되는 소프트웨어의 설치 및 사용 (2) Linux 계산서버의 부재 (3) 대용량 파일의 저장 및 관리의 3가지 문제점으로 요약할 수가 있다. 일반적으로 NGS 데이터는 보통 Exome은 10GB, Genome은 100GB이며, 적절한 장비를 보유하고 있더라도 분석에 필요한 시간은 Genome의 경우 일주일 정도 소요가 된다. 대부분 GATK (Genome Analysis Toolkit, https://gatk.broadinstitute.org)의 권고사항 (Best Practices)을 사용하며 WDL (The Workflow Description Language)로 개발된 Workflow를 다운로드 받아서 Cromwell (https://github.com/broadinstitute/cromwell)로 구동이 가능하다.
Broad Institute에서 개발한 Terra (https://terra.bio)는 클라우드를 Backend로 웹기반의 Jupyter Notebook (https://jupyter.org) 인터페이스에서 구동할 수가 있다. 기본적으로 Python 언어 (https://python.org)를 사용하여 GATK의 권고사항에 따른 정보분석 과정을 수행한다. 일주일 정도 소요되는 분석 시간은 실제 유전체진단 등 활용에 지연이 될 수 있으며, 최근에는 Illumina 社에서는 FPGA (Field-Programmable Gate Array Technology)를 사용한 DRAGEN Bio-IT Platform을 판매하고 있으며 몇 시간 이내에 분석을 완료할 수가 있다. NVIDIA 社에서는 GPU (Graphics Processing Unit)를 사용할 수 있는 Clara Parabricks (https://developer.nvidia.com/clara-parabricks) 라이브러리를 제공하며, 다양한 오믹스 데이터 분석에 필요한 기본 소프트웨어 및 Deep Learning을 이용한 인공지능 소프트웨어를 제공한다.
국내에서는 대부분 Microsoft 社의 Windows를 기본 OS (Operating System)으로 사용하고 있다. Apple 社의 macOS는 Linux 기반의 소프트웨어를 컴파일해서 사용할 수 있는 개발환경을 제공하지만, Windows 환경에서는 매우 제한적이다. 최근에 Microsoft 社는 Linux를 Kernel에 통합할 수 있는 WSL2 (Windows Subsystem for Linux 2, https://docs.microsoft.com/en-us/windows/wsl/install-win10)를 배포하였다. https://wsldl-pg.github.io/docs에서 다양한 Linux 버전에 대한 WSL2를 다운로드 받아서 사용할 수 있다. Linux는 크게 Red Hat과 Ubuntu 계열로 구분되며 시스템 관리 명령어가 다르다.
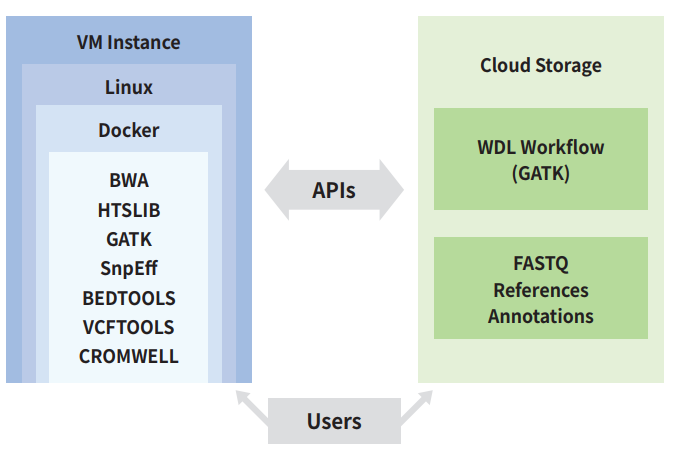
그러나, Linux를 사용환경을 구축하더라도 NGS데이터 분석에 사용되는 소프트웨어의 설치는 더 어렵다. BWA (http://bio-bwa.sourceforge.net), SAMTOOLS /BCFTOOLS/HTSLIB (https://www.htslib.org), GATK, SnpEff (http://pcingola.github.io/SnpEff) 또는 ANNOVAR (https://annovar.openbioinfor matics.org) 가 사용되며 추가적으로 BEDTOOLS, VCFTOOLS, CROMWELL 등이 필요하다. 소프트웨어의 설치 이외에 다양한 옵션을 모두 이해하기는 매우 어렵다. 모든 클라우드는 사용자가 요청한 자원 (vCPU, vMEM, DISK)을 Docker (https://hub.docker.com)를 OS로 제공한다. 일반적인 계산서버가 직접적으로 연결된 하드웨어 자원을 활용하는 반면, Docker는 OS를 image 파일로 저장하고 바로 구동한다. Docker는 모든 OS에 설치가 되며, 플랫폼에 독립적으로 docker image를 구동 (instance 생성) 할 수가 있다. 그렇다면, 여기에 NGS 데이터 분석에 필요한 모든 소프트웨어를 미리 설치하고 저장이 가능하다. https://hub.docker.com 에서 GATK를 검색해 보면, 많은 사람들이 다양한 Linux를 사용하여 정보분석 용으로 제작된 공개 image를 다운로드 받아서 사용할 수가 있다. Broad Institute의 Terra 플랫폼도 docker image를 사용한다.

정보분석 단계를 하나의 묶음으로 연결한 것을 Pipeline이라고 부른다. GATK에서 제공하는 권고사항도 하나의 Pipeline이라고 할 수가 있다. 이전에는 Shell Script로 명령어들을 하나의 텍스트 파일로 묶어서 사용하였다. 다양한 옵션들을 미리 정해 놓고 샘플, 코호트의 이름을 변수로 넘겨주고 실행하면 정보분석이 오류가 없이 완료될 때까지 기다리기만 하면 된다. 하지만, 이러한 방식은 클라우드에서 사용하기에는 분산컴퓨팅 환경에 맞지 않아 불편한 점이 있다. 최근에는 이보다 한단계 향상된 WDL 언어로 개발된 Workflow를 사용한다. GATK도 모든 권고사항을 WDL로 배포하고 있다. GATK3의 Apache 2.0 라이선스에서 GATK4의 BSD-3 라이선스로 변경이 되면서, 전체 분석 절차에 대한 자세한 설명 대신 INPUT 파일을 위한 JSON 포맷 텍스트 파일과 WDL 파일로 전체 Workflow를 제공하고 있다. CROMWELL로 구동되는 WDL은 분산컴퓨팅 환경에 적합하며, 필요한 vCPU, vMEM 및 interval (chromosome)에 의한 동시 작업 개수를 제한하여 구동할 수 있는 장점이 있다. 물론 계산서버가 구비되어 있을 경우에는 docker 이미지와 WDL Worlfkow를 사용하여 NGS데이터 분석을 수행할 수가 있다. JSON 포맷의 INPUT 파일에서 어떤 데이터를 분석할 것인지 파일의 위치를 입력해주고 계산을 진행하면 된다.
Linux가 구동되는 계산서버 환경을 어떻게 준비할 것인가? 클라우드를 활용하면 구매 비용으로 수천만원이 드는 계산서버 환경을 마우스 클릭으로 필요한 시간만큼 요금을 지불하고 사용할 수가 있다. 이러한 환경은 Amazon, Microsoft, Google 등 많은 업체에서 제공하고 있다. 예를 들어, Google Cloud (https://cloud.google.com)에서는 콘솔로 들어가서 3가지 환경을 설정한다.
첫번째로 vCPU와 vMEM의 할당을 위한 Compute Engine을 제공하고 있다. VM instance를 생성할 때 주의할 점은 데이터의 저장 보다 인터넷 전송 비용이 더 비싸다는 점이다. 따라서, VM instance는 해외에 생성하지 않고 Region/Zone을 살펴보고 서울 및 Asia 지역으로 한정해야 한다. 그렇지 않으면 각종 파일의 전송에 매우 비싼 요금이 청구될 수가 있다. Exome 및 Genome의 분석에는 매우 고사양 환경이 필요하지 않으므로 실제적으로 vCPU=8, vMEM=30 수준이면 적은 수의 샘플을 분석하기에 적합하다. 클라우드의 장점은 이러한 vCPU/vMEM 자원을 동시에 여러 개 요청이 가능하다는 점이다. 따라서 1개의 데이터 분석에 5일이 소요되면, 10개의 데이터 분석도 5일이 소요된다. 모든 작업을 동시에 자원을 할당 요청하여 진행하기만 하면 된다.
두번째로, 파일을 저장하기 위한 공간이 필요하다. Google Storage (Bucket)은 gs://로 시작하는 URI를 제공하며 Class에 따라 요금은 조금씩 다르지만 Google Drive와 달리 사용하는 만큼만 지불하면 되는 장점이 있다. 단, Google Drive와 Google Storage는 서로 파일을 이동할 수 있는 API가 없다. NGS 데이터 분석은 Compute Engine에서 VM instance를 생성하여 vCPU/vMEM을 할당 받고, 필요한 모든 파일을 미리 Google Storage에 올려 놓은 다음, 분석이 시작되면 이 파일을 복사한다. 모든 데이터 분석 과정이 끝나면 다시 Google Storage로 어떤 OUTPUT 파일들을 복사할지를 지정하면 된다.
세번째로 Container Registry는 docker image를 위한 파일의 보관 및 URI를 제공한다. 사전에 NGS데이터 분석을 위한 모든 소프트웨어를 탑재한 docker image를 Container Registry에 업로드하면 VM instance에서 URI를 통하여 docker image를 불러와서 실행할 수가 있다. 여기까지 설명한 Google Cloud에서 분석 환경은 API를 통하여 좀더 쉽게 해결이 되는데 APIs & Services 메뉴에 들어가면 lifesciences API를 검색하여 활성화하고 사용할 수가 있다. API는 자원의 할당, Google Storage에서 파일의 복사, 분석 Workflow의 실행, OUTPUT 파일의 복사, 할당된 자원의 반납 등 전반적인 과정을 대행한다.
현재 수백만명의 인간유전체에 대한 NGS데이터가 생산되었고, 전세계적으로 수십 페타바이트 (Petabyte = 1000 Terabyte) 분량이라고 예상할 수가 있다. 국외에 있는 파일을 국내로 전송하기도 매우 어렵고 그 많은 데이터를 저장할 수 있는 스토리지서버를 갖춘 기관도 국내에 많지 않다. 개인연구자가 접근하기에는 불가능하다. 원자자의 허락이 없이는 데이터에 대하여 접근할 수 없을 뿐만 아니라, 그 많은 데이터를 직접 다운로드 받아서 원하는 정보분석을 수행하고 결과를 얻는 것은 불가능하다고 생각할 수 있다. 이러한 문제점에 대한 해결을 위하여 암유전체, UK Biobank, 의료영상이미지 정보들은 모두 클라우드 환경에서 공유가 된다. NGS 데이터 정보분석을 위해서 클라우드를 사용하는 것이 일반화된다는 의미가 된다.
마지막으로 본문에서 언급한 Google Cloud 환경에서 Exome 및 Genome 데이터 분석 WDL workflow는 http://dnaseq.exome.kr 및 http://rnaseq.exome.kr 에서 간단한 정보 입력후 다운로드가 가능하다.