Nortable Research
Scalable single-cell profiling of chromatin modifications with sciCUT&Tag
Technology Trend 학회뉴스씨젠의료재단
Cleavage under targets and tagmentation (CUT&Tag) 기술은 면역침전법을 사용한 검사를 빠르게 대체하고 있다. 저자들은 CUT&Tag 기술에 바코드법을 결부시켜 고해상도와 대량 처리가 가능한 sciCUT&Tag (single-cell combinatorial indexing CUT&Tag) 기술을 개발하여 Nature Protocol에 게재하였기에 소개하고자 한다.
CUT&Tag 법은 Tn5 cut and-paste transposase fused to a protein A 잔기(pA–Tn5)를 이용하여 genomic loci에 항체표적을 설정하여 PCR용 어댑터로 태그 하는 검사법으로, 이를 변형한 여러 가지 방법이 발표되어 왔다. 저자들은 이전에 발표했던 CUT&Tag 법을 개선하여 sciCUT&Tag 법을 새로 발표하였다.
자세한 내용은 첨부된 원문을 참고하기 바라며, sciCUT&Tag 실험 개요를 간단히 설명하면 다음과 같다( 그림1 참조). 먼저 세포를 1차 항체로 처리하여 배양하고 washing 후 2차 항체로 처리하여 재배양한다. 96 웰 플레이트에 분주한 후 pA-Tn5로 태깅한다. 96웰 검체를 풀링하여 TaKaRa ICELL8 장비로 5,184웰 칩에 재분주하고 PCR 증폭을 시행하여 2차 바코드(여러 가지 색의 선분 으로 표시됨)를 태깅한다. 서로 다른 검체에서 유래한 세포 들은 각각 파랑, 노랑, 빨강으로 나타내었다. 실험이 끝나면 sciCUT&Tag 데이터를 처리한다(그림 1-b). 한 웰에서 여러 가지 SNP 바코드를 가진 유전형들이 검출될 경우 collision removal 시행하여 noise를 제거한다. 한 검체 내에서 세포아형들을 식별하여 도표기반 클러스터링으로 표시( 차원 축소)한다. 처리된 세포아형들은 특정 세포 유형으로 지정된다.
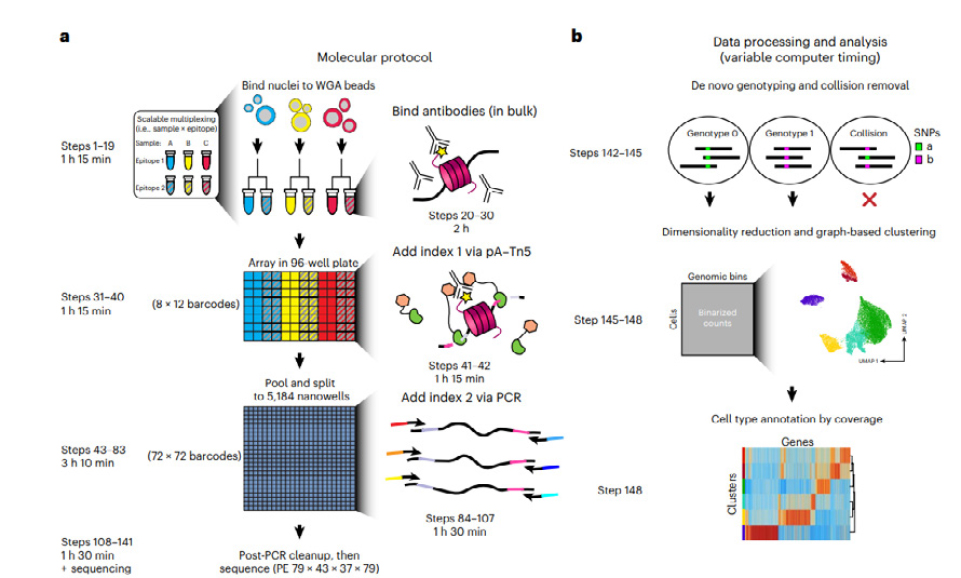 <그림 1> sciCUT&Tag 실험 작업흐름 개요.
<그림 1> sciCUT&Tag 실험 작업흐름 개요.
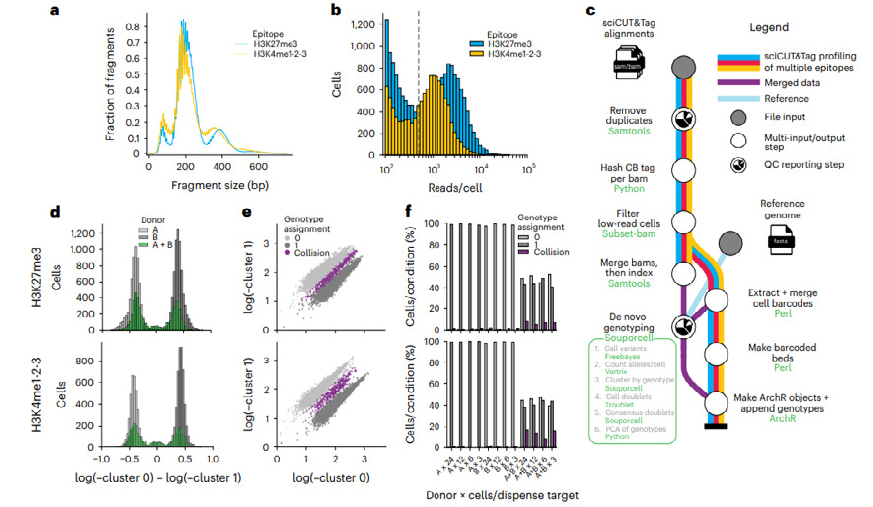 <그림 2> sciCUT&Tag에서 SNP 기반 collision removal에 의한 유전형 분석법 검증
<그림 2> sciCUT&Tag에서 SNP 기반 collision removal에 의한 유전형 분석법 검증
특기할 점은, nanowell dispenser를 활용하여 96웰 플레이트에서 Tn5 태깅을 시행한 핵들을 재부유시켜 5,184 나노웰에 재분주, PCR 시행하여 서열분석용 라이브러리를 제작하였으며, 이러한 nanowell dispenser를 사용함으 로써 처리량과 분주 정확도(웰 당 12-24 개의 핵 분주)-개별 세포 프로파일 정확도-를 획기적으로 상승시켰다는 것 이다. 또한 이로 인해 데이터 분석 단계에서 collision removal 적용을 용이하게 하여 크로마틴 에피토프 프로파일 링을 정확하고 효율적으로 시행할 수 있었다.
혼합 PBMC를 이용하여 시행한 sciCUT&Tag에서 SNP 기반 collision removal에 의한 유전형 분석법을 검증하 였다(그림 2 참조). 그림 2-a에 H3K27me3(파란색)과 H3K4me1-2-3(노란색)의 조각 크기 분포가 표시되었다. 500 reads 이하의 바코드는 분석에서 제외된다. 그림 2-c 는 SNP 기반 collision removal로 유전형분석을 시행하기 위해 Souporcell24에 구축된 Nextflow26 파이프라인 모식도를 나타낸다. H3K27me3(위)와 H3K4me1-2-3( 아래)의 검체별 유전형 할당 클러스터 확률에 대한 세포수를 2-d에 나타내었고, 2-e에서 클러스터 확률에 따라 할당된 두 가지 유전형을 서로 다른 색상으로 표현하였다. 기증자 세포와 타겟 유전형을 각각 검사한 것과 혼합하여 검사한 경우 collision removal이 일어나는 빈도를 2-f에 표시하였으며, 단일 유전형 검체를 검사한 경우 collision removal이 거의 일어나지 않고, 혼합검체의 경우 collision removal이 유의하게 높이 발생하여 효과적으로 유전형을 구분할 수 있음을 검증하였다.
sciCUT&Tag을 여타 single-cell CUT&Tag법에 비교하 였을 때 다음과 같은 장점이 있다: 첫째, 처리속도가 여타 single-cell CUT&Tag 법에 비해 최소 4배 이상 빨라졌다 (전 과정이 1.5일에 완료). 둘째, 로딩조건을 최적화하였을 경우, 칩당 최대 4만 개의 세포를 로딩하여, 라이브러리 프렙과 서열분석 비용을 세포 당 0.11 달러까지 줄일 수 있어 높은 가성비를 자랑한다. 셋째, 동일한 풀에 있는 핵들을 여러 번 반복 분석하여 single cell profile을 더 특이적으로 검출할 수 있다. 넷째, 비용 절감과 더불어 해상도가 증가 하였다.
다만, 저자들이 sciCUT&Tag법에서 약속한 높은 성과를 위해서는 장비-특히 nanowell dispenser (여기서는 TaKaRa ICELL8의 nanowell dispenser)-가 상당히 중요 함을 알 수 있는데 해당 장비의 가격을 알 수는 없었지만, 0.11 달러라는 비용은 조금 많이 과장된 것으로 생각된다. 하지만 sciCUT&Tag법이 기존 검사법들에 비해 많은 이점을 가졌기에, 비교유전체 분석과 드문 세포형(形) 혹은 세포의 상태를 규명할 때 보다 넓은 범용성을 가진 검사법으로서 이용될 수 있을 것으로 생각된다.
[References]
1. Janssens, D.H., Greene, J.E., Wu, S.J. et al. Scalable single-cell profiling of chromatin modifications with sci-CUT&Tag. Nat Protoc (2023). https://doi.org/10.1038/s41596-023-00905-9

