Nortable Research
Application of CRISPR/Cas9-based mutant
enrichment technique to improve the clinical
sensitivity of plasma EGFR testing in patients with
non-small cell lung cancer
Technology Trend
학회뉴스
분당차병원 진단검사의학과과
서론
Epidermal growth factor receptor (EGFR) 내성 변이를 민감하게 검출하는 것은, 1차EGFR-tyrosine kinase inhibitors (TKI) 투약 후 병변이 진행된 비소세포폐암 환자에서
2차 치료제로 3세대 TKI를 선택하는데 도움이 됩니다. 1차 EGFR-TKI 요법에 대한 내성의 약 50~60%는 후천성 p.Thr790Met (T790M) 변이에 의해 발생합니다. 일반적으로
≥10 copies/mL의 T790M은 현재 이용 가능한 방법으로 검출할 수 있지만, 환자의 약 50%는 T790M의 copy수가 낮아(<10 copies/mL) 검출하기 어렵습니다. 그럼에도 불구하고
낮은 T790M copy수 (<10 copies/mL)를 가진 환자는 3세대 EGFR-TKI에 대해 높은 T790M copy수(≥10 copies/mL)를 가진 환자와 비슷한 반응을 보였습니다.
비소세포폐암 환자의 경우, 순환 종양 DNA(circulating tumor DNA)를 검출하기 위한 액체 생검이 가장 일반적으로 시행되었습니다. FDA의 승인을 받은 Roche cobas® EGFR Mutation Test v2 (Roche Molecular Systems, Pleasanton, CA, USA)와 같은 실시간 PCR(qPCR)은 사용이 간편하고 상대적으로 저렴하기 때문에 임상 검사에서 널리 사용됩니다. 그러나 이 검사법은 변이의 검출을 위해 특정 EGFR 변이의 최소 100 copies/mL가 필요합니다. EGFR 변이의 대립 유전자 빈도가 0.1% 미만이면 qPCR로 거의 검출할 수 없습니다. 많은 연구자들은 ddPCR (droplet digital PCR) 및 NGS (next-generation sequencing)를 사용하여 cfDNA (cell-free DNA) 변이를 민감하게 감지할 수 있다고 제안합니다. 그러나 대립유전자 빈도가 0.1% 미만인 변이는 현재 기술로는 일관적으로 검출할 수 없습니다. 또한, 고도로 단편화되고 소량으로 존재하는 ctDNA, 야생형 (WT) 대립 유전자의 높은 백그라운드 및 cfDNA의 빠른 제거는 cfDNA에서 특히 낮은 대립 유전자 빈도 변이를 검출하는 데 장애가 됩니다.
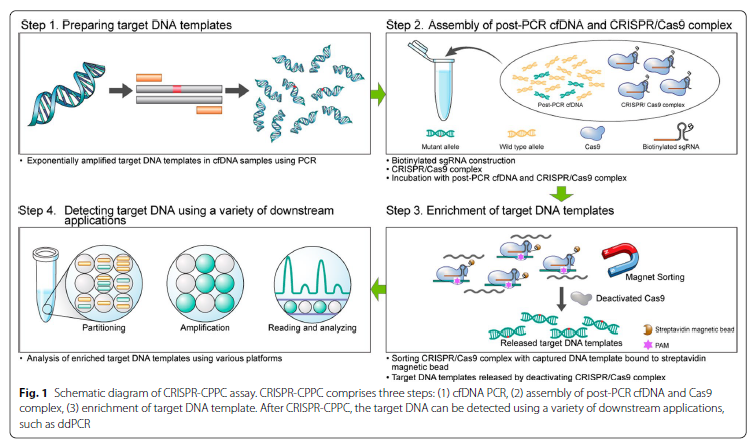
Clustered regular interspaced short palindromic repeats(CRISPR)/CRISPR-associated protein 9 (Cas9) 시스템은 검출 능력을 향상시키기 위해 분자 진단 분야에 도입되었습니다. 활성 CRISPR/Cas9은 유전자 편집 및 표적화를 위한 다재다능하고 정확한 도구입니다. 본 연구에서 저자들은CRISPR system combined with post-PCR cfDNA (CRISPR-CPPC)로 불리는 새로운 변이 강화(mutation enrichment) 기술을 제안합니다.
방법
본 연구에서는 변이 대립 유전자의 참조 표준과EGFR-TKI 치료 중 또는 치료 후에 임상적으로 진행된 non-small cell lung cancer (NSCLC) 환자의 cfDNA로 CRISPR-CPPC
분석을 검증했습니다. EGFR T790M 검출의 분석적 성능은 CRISPR-CPPC assay 결과와 qPCR 또는 ddPCR 결과를 비교하여 평가하였습니다. CRISPR-CPPC는 (1) cfDNA PCR,
(2) post-PCR cfDNA와 CRISPR/Cas9 복합체의 조립, (3) 표적 DNA 주형의 강화의 세단계로 구성됩니다. CRISPR-CPPC 후 다양한 검사법을 사용하여 표적 DNA를 검출할 수 있습니다.
이 연구에서는 T790M을 검출하기 위한 방법으로ddPCR을 사용했습니다. 실험 및 검증의 세부사항은 원문 [1]을 참고바랍니다.
Roche cobas® EGFR Mutation Test v2를 사용하여 EGFR 유전자 변이 검사가 필요한 51명의 환자로부터 총 60개의 샘플을 수집했습니다. 1세대 또는 2세대 EGFR-TKI 치료 중 또는 치료 이후에 EGFR 변이가 있는 NSCLC 환자만 포함하였습니다.
결과
총 21개 논문을 대상으로 분석을 시행하였으며, 하위 집단에 따라 상이한 결과를 보였습니다. 민감도는 나라와 출판 년도에 따라 상당한 차이를 보였으나 임상적으로 유의미한
수준은 아니었습니다. 전반적인 민감도는 98.83%, 특이도는 99.93%로 높았으며 NPV와 정확도 또한 각각 100%, 99.91%로 높은 소견이었습니다. 하지만 PPV는 T21에 대한
NIPS의 PPV에 비해 확연히 낮은 결과를 보였습니다.
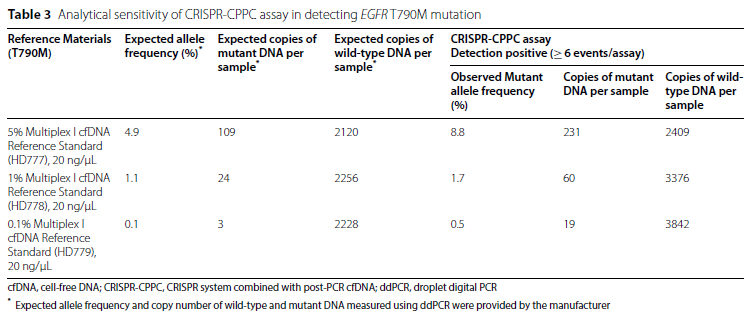
두 가지 이상의 실험 방법(cfDNA, 조직 또는 다른 유형의 샘플, NGS, ddPCR 및 CRISPR-CPPC의 qPCR)을 통해 T790M에 대해 양성으로 테스트된 샘플은 진양성으로 간주했습니다(표 S3). 다중 분석 결과에 따르면 CRISPR-CPPC 분석과 ddPCR의 민감도는 각각 92.0%와 64.0%였습니다.
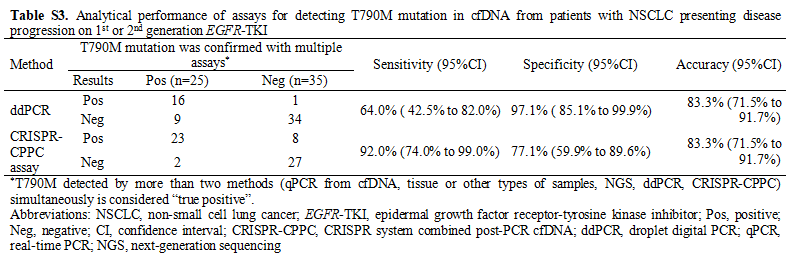
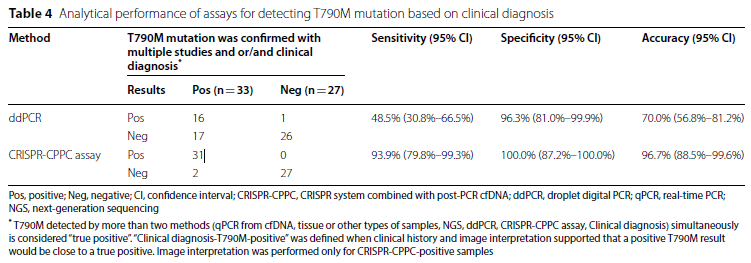
임상 진행의 최종 임상 진단은 환자의 병력 및 방사선 소견의 통합을 기반으로 종양 전문의에 의해 이루어졌습니다. 연구진은 최종 임상 진단을 포함하여 시험 대상자의 의료 기록을 후향적으로 검토했습니다. 표 4는 다중 분석 및 최종 임상 진단 결과를 기반으로 한 CRIPSR-CPPC 분석 및 ddPCR의 분석 성능을 나타냅니다. CRISPRCPPC 분석의 민감도와 특이도는 각각 93.9%와 100.0%까지 증가했습니다.
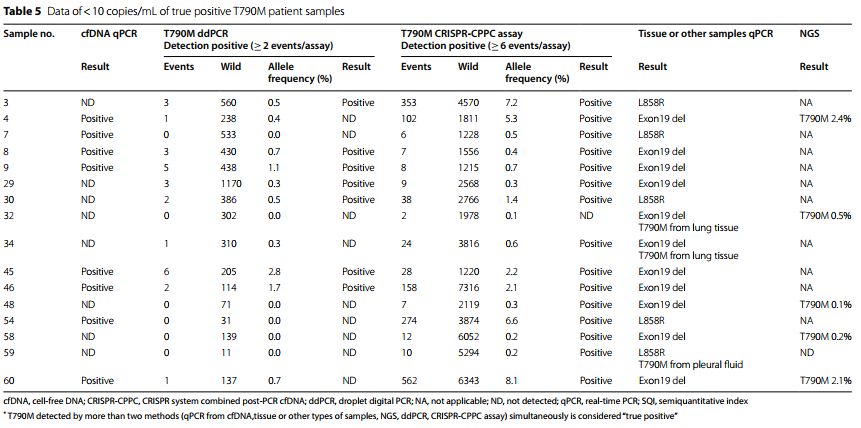
<10 copies/mL 인 “진양성”의 T790M cfDNA를 CRISPR-CPPC 방법을 이용하여 분석하였을 때 양성률 93.8%(15/16)로 ddPCR (43.8%(7/16))에 비해 높았습니다(표 5). CRISPR-CPPC 분석은 특히 ddPCR에 의해 T790M이 낮은 copy수 또는 T790M 음성인 샘플에서 T790M을 검출하는 향상된 감도를 보여주었습니다.
논의
CRISPR/CAS9 시스템은 낮은빈도로 존재하는 변이 DNA를 감지하기 위해 개발되어 적용되었습니다. 따라서, 본 연구에서는 낮은 copy수의 T790M 변이 (<10 copies/mL)의 경우
진단 민감도를 높이기 위해 추출 단계 후 변이 대립 유전자를 풍부하게 할 수 있는 CRISPR-CPPC 분석을 개발하고 검증했습니다. CRISPR-CPPC는 몇 가지 장점을 보여주었습니다.
첫째, primer만 설계한다면 사용하기 쉽습니다. 둘째, 낮은 copy수의 변이를 성공적으로 강화하였습니다. 셋째, 이전에 borderline(경계성의) 결과를 명확히 할 수 있었습니다.
째, 실험에 기초하여, enrichment reaction은 동일한 양 (약 0.4 ng)의 post-PCR cfDNA로 수행 될 수 있으며, 이는 CRISPR-CPPC가 표준화 된 공정이 될 수 있음을
나타냅니다. 마지막으로, CRISPR-CPPC는 cfDNA에 대한 PCR 단계를 추가함으로써 DNA 주형 손실을 보정 하였습니다. 그러나 DNA 손실에 대한 이 보정 단계는 오염 가능성이
있으므로 신중한 취급이 필요합니다.
이 연구는 CRISPR- CPPC 분석법의 성능이 현재 이용 가능한 다른 방법의 성능보다 훨씬 우수하며 임상 환경에서 쉽게 사용할 수 있음을 보여주었습니다. 따라서, CRISPR-CPPC는 유전자 발현 프로파일링, 진단 및 적절한 치료 요법의 선택을 용이하게 하기 위해 임상적으로 적용될 수 있습니다.
[참고 문헌]
[1] Kim, B.; Kim, Y.; Shin, S.; Lee, S.-T.; Cho, J.Y.; Lee, K.-A. Application of CRISPR/Cas9-based mutant enrichment technique to improve the clinical
sensitivity of plasma EGFR testing in patients with non-small cell lung cancer. Cancer Cell Int. 2022, 22, 1–11.

