GC Genome
암은 초기단계에서는 대부분 뚜렷한 증상이 없기 때문에, 적절한 스크리닝 검사가 이루어지지 않으면 더 늦게 발견된다. 한국에서도 현재 주요 6대 암 검진 프로그램이 국가에서
제공되고 있지만, 이러한 프로그램은 특정 암종에 중점을 두고 있으며, 검진 방법의 성능에도 한계가 존재한다. 이에 다중암을 조기에 발견할 수 있는 혁신적인 기술이 개발되고
도입된다면 암 환자의 치료기회를 높이고, 생존율을 향상시킬 수 있을 것으로 기대된다.
액체생검은 혈액에 떠돌아다니는 종양 DNA 또는 RNA를 활용해서 암을 진단하는 방법이다. 3-4기의 이미 진단 받은 암 환자에서 표적항암제를 선정하기 위한 용도로 주로 활용되고 있다.
1-2기의 초기암에 적용되지 못하는 이유는 종양에서 유래된 ctDNA가 전체 cfDNA중에서 아주 소량 존재하기 때문에, 기존 돌연변이에 기반한 검사법으로는 높은 민감도를 달성하기
어렵기 때문이다.
액체생검의 민감도의 한계를 극복하고 암의 조기 진단에 이용하기 위해서 여러 회사들이 메틸레이션 (Methylation) 마커의 활용과 cfDNA의 단편체학 (fragmentomcis)을 활용하고 있다.
다중암 조기진단분야에서 선두주자인 그레일(Grail) 사는 cfDNA의 메틸레이션 마커들의 조합으로 50 여종이상의 암을 진단하고 분류하고 있으며, 이미 2021년부터 검사서비스를 제공하고 있다. 특히 영국의 NHS는 공동연구를 통하여 14만명의 데이터를 확보하고 있으며, 향후 백만명이상으로 확대할 계획을 가지고 있다. 이그젝트 사이언스(Exact Sciences) 사는 약 만여명의 여성을 대상으로 기존의 검진검사와 비교하는 Detect-A 연구를 발표한 바 있다 (1). DELFI Diagnostics사는 cf-WGS에 기반한 DELFI 알고리즘을 개발하였으며, 폐암 및 간암등 다양한 암에서 추가 임상 연구를 진행중이다 (2)
GC Genome에서는 다중암의 조기진단을 목표로 KAIST와 공동으로 인공지능 알고리즘을 개발하 였으며, 이를 올해 Nature communication지에 “Integrative modeling of tumor genomes and epigenomes for enhanced cancer diagnosis by cell-free DNA” 라는 제목으로 발표한 바 있다. (3).
이 연구는 9가지 암 종류의 2,125명의 환자 샘플과 1,241명의 정상 대조 샘플에서 cf-DNA WGS을 수행하였으며, 암환자를 검출할 수 있는 인공지능 알고 리즘을 개발하였고, 추가로 208명의 암샘플과 214 명의 정상 대조군 샘플에서 성능을 검증하였다. 암환자를 초기에 민감하게 검출하기 위해서 genome model과 epigenome model를 개발하여 이를 통합 하였다(Fig 1). Genome 모델은 저밀도 전장유전체 에서 생산된 변이들의 밀도와 signature를 인공지능 으로 학습시켜 암을 진단하고 분류하고자 하였다. 이미 암종마다 돌연변이 빈도와 signature가 다르게 나타난다는 것은 알려져 있다. 알고리즘 개발을 위해서 PCAWG에서 확보된 데이터셋에서 암종류별로 high mutation density와 low mutation density 영역을 선별하였고, 150개의 변이유형을 모델의 피처로 사용하였다. 또한 저밀도 전장유전체 시퀀싱(low depth cfDNA-WGS)의 경우 많은 시그널 노이즈를 가지고 있기 때문에 20,529명의 건강한 샘플들을 바탕으로 각 샘플마다 변이 필터링을 수행하였다. 변이 필터링을 통해서 정제된 데이터를 2726개의 유전체 영역에서의 변이 밀도와 150개의 변이 유형을 구하 고, 인공지능 알고리즘으로 학습하여 암 환자와 정상 인을 감별할 수 있는 알고리즘을 개발하였고, MGI와 Illumina로 학습 데이터에 대해서 AUC (Area Under the ROC Curve) 0.9 이상의 우수한 성능 달성하였 다. (표 1) epigenome 모델은 NDR(NucleosomeDepleted Region)이 암 환자와 정상인에서 또한 암종마다 다른 패턴을 보인다는 것을 이용하였다.
Epigenome 모델의 프로필을 구축하기 위해서 TCGA 샘플의 ATAC-seq 데이터를 활용하여 영역을 선별하고 합성곱 신경망을 이용하여 모델을 구축하였다. 여러 데이터셋에서 Genome 모델과 Epigenome 모델을 결합하였을 때 가장 우수한 성능을 확인할 수 있었다. (Fig 1)
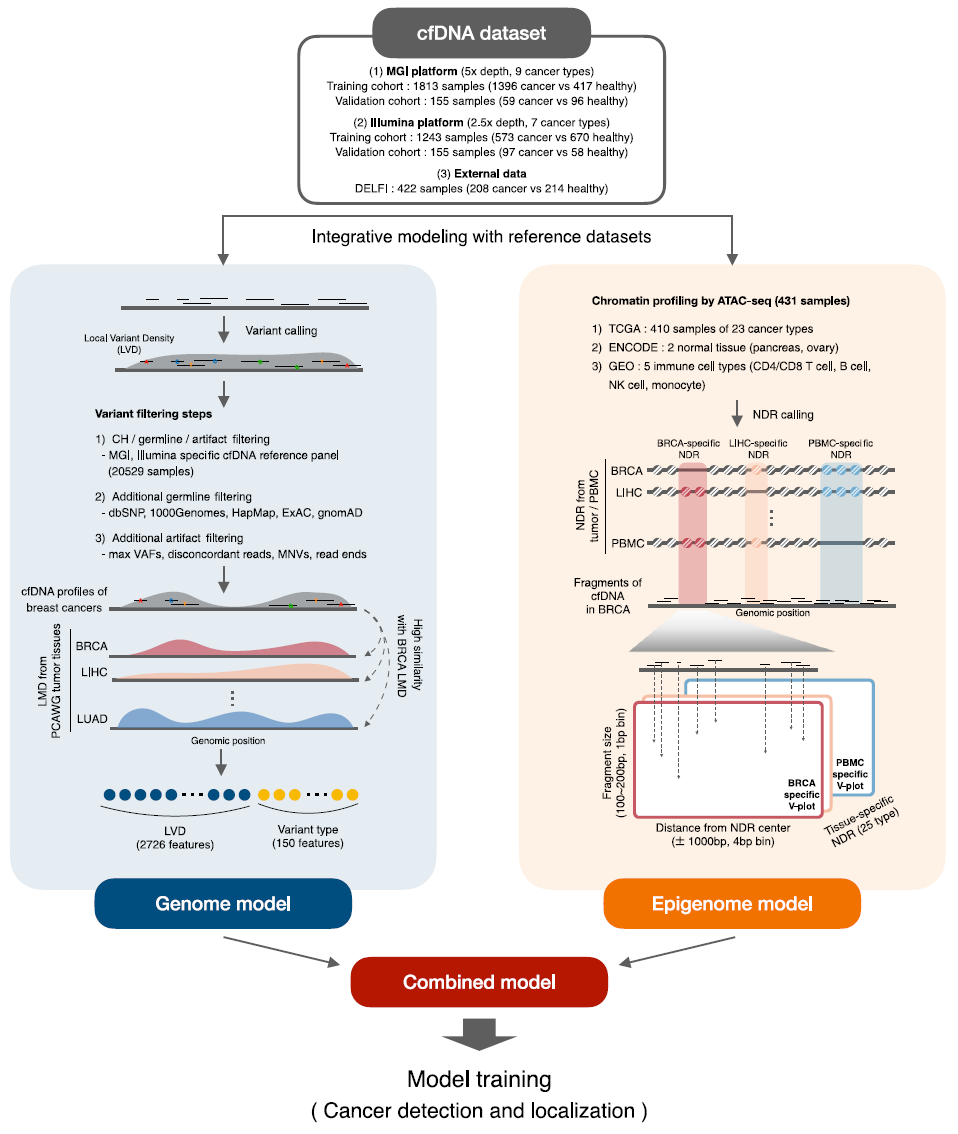 Fig 1. genome model과 epigenome model
Fig 1. genome model과 epigenome model
GC Genome에서는 이러한 결과를 바탕으로 다중암 조기진단의 가능성을 확인 하였으며, 추가적인 임상 연구를 진행중이며 상용화 준비중에 있다. 이러한 결과 들이 암 스크리닝 검사의 패러다임을 변화시킬 것으로 혁신적인 기술이 될 것으로 기대한다.
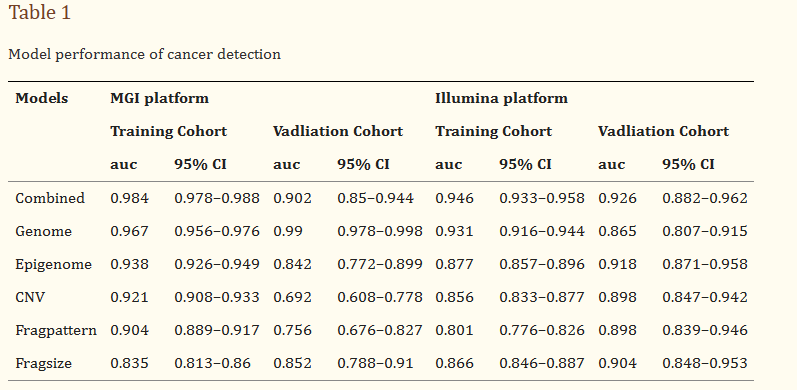 표 1. Model performance of cancer detection
표 1. Model performance of cancer detection
[참고 문헌]
1. Lennon AM, Buchanan AH, Kinde I, et al. Feasibility of blood testing combined with PET-CT to screen for cancer and guide intervention. Science. 2020 Jul 3;369(6499):eabb9601
2. Cristiano, S., Leal, A., Phallen, J. et al. Genome-wide cell-free DNA fragmentation in patients with cancer. Nature 570, 385–389 (2019)
3. Bae M, Kim G, Lee TR, et al. Integrative modeling of tumor genomes and epigenomes for enhanced cancer diagnosis by cell-free DNA. Nat Commun. 2023 Apr 10;14(1):2017. doi: 10.1038/s41467-023-37768-3.

