삼성서울병원
대규모 유전체 염기분석사업인 [국가바이오빅데이터 사업] 에서 희귀질환 시범 사업이 2020년 6월부터 시작되었다. 본사업인 100만명의 유전체 염기분석사업이 진행된다면 국내 정부주도 생물 의료 (biomedical) 연구 개발에서 전례 없는 대규모 투자와 규모가 될 것이다. 이는 미국, 영국 등 외국에서 진행하는 사업에 대응하는 성격이 크다. 이 사업의 의미를 인간유전학 발전의 맥락에서 검토하고, 현행 사업의 진행에 대해 설명하고자 한다.
1. 인간유전학의 발전 A. 단일유전자 (희귀) 질환의 유전자 분리: Linkage analysis 인간 유전학의 가장 큰 실용적 관심은 질환과 유전자의 관계이다. 생화학적 지식과 효소의 정제를 통한 페닐케톤뇨증의 원인유전자인 PAH 유전자의 분리나, 세포유전 분석을 통해 분리한 Duchenne Muscular Dystrophy 의 DMD 유전자 같은 개별유전자의 연구 성과나 특성을 바탕으로 한 원인유전자의 규명은 시간과 노력이 많이 드는 매우 고단한 일이었다. 1983년 James Gusella 박사는 linkage analysis를 통해 헌팅턴 병 유전자가 4p염색체에 있는 것을 증명하였다. 이어서 1989년 F. Collin 박사와 LC. Tsui 박사가 Linkage analysis를 이용하여 낭성 섬유종 (cystic fibrosis)의 CF 유전자를 분리하였다. 원인유전자에 대한 지식이 전혀 없어도 유전질환 환자들의 가족들 검체를 모아서 linkage analysis를 한다면 질환 유전자를 규명할 수 있다는 것은 획기적인 발전이었다. 따라서, 유전질환 가족들을 모아서 유전자 분리를 하는 것이 1990년대 인간유전연구의 중심 연구 방법이 되었다. 따라서 1990년에 시작한 인간유전체 사업(human genome project) 에서 가장 먼저 시작한 일은 특정 염색체의 유전자들의 염기서열 분석이 아니라 전체 염색체들에 퍼져 있는 다형성 표지자에 대한 정확한 지도를 만드는 것이었다. 이들을 이용하여 2000년까지 유전성 유방암 유전자인 BRCA1, BRCA2, 윌슨병 유전자인 ATP7B, 신경섬유종 유전자인 NF1, NF2 등 대략 1000개 정도의 단일유전자 (희귀) 질환의 원인 유전자가 규명되었다. 필자가 2003년에 발견한 유전성 말초신경병증 환자의 가족으로부터 2005년에 CMTX5 (Charcot-Marie-Tooth disease X5)를 정의하고 2008년에 원인유전자를 보고한 것도 인간유전체 사업에서 만들어진 linkage map 정보가 없었으면 불가능한 일이었다.
B. 복합 (complex) 질환의 유전자 분리: from linkage analysis to association analysis 처음 단일유전자 질환의 성공을 바탕으로 당뇨, 조현병 등 복합질환(complex disease)으로 연구대상이 확대되었다. 그러나, 이들의 연구는 단일(희귀)질환에 비해 뚜렷한 결과를 보여주지 못했다. 1996년 Risch박사와 Merikangas박사는 linkage analysis를 이용한 복합질환의 원인유전자 발견은 현실적으로 불가능하며 association analysis 가 대안임을 simulation study를 통해 주장하였다. 이들의 주장은 곧바로 학계의 인정을 받아 복합질환의 연구 중심이 association analysis로 바뀌게 되었다. 2003년 인간유전체 사업의 성공적인 종료 선언에 뒤이어 Hapmap 사업이 시작된 것은 복합질환에서 association analysis를 위한 표지자 지도작성사업이었다. 이들을 통해 단일 염기 다형성 (Single Nucleotide Polymorphism; SNP)의 지도가 만들어졌고, 이들을 이용한 Genome Wide Association Study(GWAS)가 2005년부터 시작되었다. GWAS 연구로 알게 된 사실은 복합질환은 다양한 단일유전자 질환들의 합이 아니라 보통 (modest) 정도의 유전적 경향을 가진 위험인자들의 합 혹은 상호작용으로 질환이 생긴다는 것이다. 즉, GWAS로 찾아낸 질병관련 SNP들을 검토하면, 발견된 SNP들의 90%~95%가 noncoding sequence에 위치한다. 이는 단일 유전자(희귀) 질환의 병인성 돌연변이가 주로 coding sequence에 위치하는 것과 대조적이다.
C. 질환의 유전적 구조(genetic architecture) 개념의 부상 단일유전자 질환들의 원인유전자 변이들과 복합 질환에서 GWAS 로 발견한 질환관련 이환유전자(susceptible gene)들은 전술한 바와 같이 서로 다른 특성을 가지고 있다. 또 복합질환의 범주에서는 단일유전자원인들과 이환유전자들의 누적에 따라 생기는 질환이 섞여 있다. 예를 들면, 유방암에서 BRCA1, BRCA2 등 단일 유전자 질환의 환자 군과 이환유전자들의 작용으로 유방암이 생기는 환자군들이 섞여 있다. 그런데, 단일유전자 질환의 상대적 빈도나, 개별 유전자들의 질환의 기여 방식, 유전자들의 상호 작용방식은 질환마다 다르다. 즉 알츠하이머, 심장병, 당뇨, 조현병 등 다양한 복합질환에서 질환들의 유전적 구조는 모두 다르다. 따라서 복합 질환의 경우, coding sequence와 non coding sequence를 모두 분석해야 genetic architecture를 이해할 수 있다. 즉 이것은 전장유전체 염기서열분석(whole genome sequencing; 이하 WGS)이 필요한 이유가 된다.
D. 정밀의료(precision)의 등장 질병에 대한 유전 지식이 축적됨에 따라 질환 유전자를 직접 조절하는 표적치료제들이 등장하게 되었고 부작용을 최소화하기위한 약물유전체 지식들도 축적되었다. 따라서, 개인들의 유전적 특성에 따른 맞춤 의료 혹은 정밀의료의 초기 성공 사례는 광범위한 정밀의료의 적용이 가져올 건강에 대한 획기적인 성과를 기대하게 만들었다. 뿐만 아니라40억 달러가 투자된 인간유전체 사업의 경제적 효과가 9조 6500억 달러로, 178배 투자효과가 있었다는 평가는 경제적으로도 정밀의료가 매력적인 사업이라는 것을 보여주었다. 정밀의료는 국가적으로도 투자를 할 만한 산업이 된 것이다.
2. 대규모 유전체 염기분석사업의 해외 사례 A. 미국의 사례 미국은 2000년대 초부터 산발적인 여러 프로그램이 있었으나, 2015년 “Precision Medicine Initiative” 라는 이름으로 기획하여, “All of Us” 라는 프로그램으로 2018년부터 국가적인 사업으로 진행하고 있다. 2022년 3월 현재 약 450여개 검체수집 및 측정기관에서 470,000여명의 참여자를 확보하고 있고, 344,000여개 검체가 수집되고 있으며, 참여자 상태, 약물정보, 검사기록, 시술 등 의료기록 (Electronic Health Records)과 함께 설문조사, 과거력, 생활습관, 운동 정보 등 11개 분야로 나누어 수집하고 있다. 이 사업의 특징은 “All of us”라는 말처럼 데이터 피수집자인 참여자들이 자발적으로 참여하도록 하고 있다. 기존 연구와 달리 데이터 연구자들인 경우, 우선 소속 기관이 All of us가 요구하는 규정에 동의하여 프로그램에 참여한 후에 소속기관 연구자들이 이들 data연구에 참여할 수 있도록 하고 있다. 2022년 3월 현재 1125개의 연구 프로젝트가 진행되고 있으며 진행되는 프로젝트를 인터넷에서 검색할 수 있도록 하고 있다. 2022년 3월 17일 미국 국립보건원은 약 10만명의 WGS 자료와 16만 5천명의genotyping arrary 자료를 공개하였다. 대규모 WGS 자료 기반의 유전체 분석 시대가 시작되었음을 알리는 사건이다.
B. 영국의 사례 영국은 Genomics England라는 프로그램으로 진행하고 있으며 암, 100,000 명의 WGS, 신생아 유전체 프로그램을 내용으로 하고 있다. 희귀질환의 초기 결과에서 2183명의 proband에 대해 25%의 진단율 성적을 보고 하고 있다.
3. 한국의 국가 바이오빅데이터 사업 A. 배경 미국, 영국 등 해외 유전체사업을 benchmarking 하여 다부처 연구개발사업으로 진행하고 있다. 본 사업 시행 전에 시범사업으로 희귀질환자들을 대상으로 2020년 7월부터 약 2년간 15,000명의 참여자 수집을 목표로 전국17개 의료기관이 참여하고 있다. 이들의 임상정보·유전체 데이터를 국가적 차원에서 구축하고, 희귀질환 원인 규명 및 정밀의료 활성화, 유전체에 기반한 진단 등 바이오헬스 산업 발전을 위한 국가적 차원의 바이오 빅데이터 구축 및 활용 체계 마련을 목표로 하고 있다. 이 사업은 2023년부터 시행을 계획하고 있는 100만명에 대한 선도사업 (Pilot program)으로 본 사업에 대한 토대 마련의 의미도 있다
B. 희귀질환 피수집 참여자의 조건
대상은 유전자 이상 및 유전자 관련 배경이 강력히 의심되는 희귀질환으로 판단되는 환자에서 다음 세 가지 조건 중 하나를 만족하는 경우에 해당된다.
1) 가족력이 뚜렷하게 있으면서 본인 혹은 이환된 다른 구성원 중 정확한 진단이 이루어지지 않은 경우
2) 기존 제도권 내에서 진단을 위한 1차 유전자 검사 (단일 유전자 또는 시행가능한 유전자 패널검사 등)를 시행하였으나 음성이 확인된 경우
3) 기존 검사로 확진이 되었으나 비전형적인 임상 양상을 혹은 경과를 보이는 경우
이 조건을 만족하는 경우, 환자와 환자의 부모, 이환된 형제 자매중 1인까지 최대 한 가계 내에서 4인까지 참여할 수 있다.
C. 사업의 흐름 희귀질환자가 희귀질환 협력기관을 방문해서, 참여 동의서를 작성하고 소변과 혈액을 제공하면, 이들 검체와 환자 질병관련 정보가 질병관리청으로 전달되고, 이를 염기서열분석과 함께 의심 유전자 변이가 확인되면, 이를 바탕으로 진단참고 보고서가 작성되어 보고된다. 해당 기관은 이 보고서를 참고로 하여 해당 변이 확진검사를 자체적으로 시행하여 진료에 이용하게 된다(fig. 1). 사업의 진행 1차년도 목표인 5000명 참여자 등록을 달성했고, 2차년도 연간 목표인 10,000명 등록을 진행중이다.
D. 현재까지 결과 필자는 필자가 속한 의료기관의 희귀질환 협력기관 대표자로서, 유전 클리닉을 통해 환자 참여와 검체 수집, 검사 후 유전상담, 협력기관의 진단지원위원회 책임자로서 활동하고 있고, 1차년도의 진단참고보고서 생산 책임자로서 활동 하였다. 1차년도 변이를 찾아낸 성적은 대략 16% 내외이다. 본 사업은 기존의 유전자검사에서 음성인 경우에 등록하게 됨으로 외국의 다른 연구 성적과 비교하기는 어렵다. 그 외 구체적 성적이나 통계 또는 유전 연구 측면에서는 질병관리청 발표 자료나, 많은 다른 기회가 있을 것으로 생각한다. 여기에서는 연구자가 아닌 희귀질환의 유전 진단자의 입장에서 필자가 느낀 경험과 전망에 대해 이야기하고자 한다.
1. 현재 유전 분석기술은 WGS를 하더라도 모든 유전적 원인을 찾을 수 있을 만큼 발전한 것은 아니라는 것이다. 분명히 단일 유전자 질환 가계로 의심되더라도 WGS에서 찾지 못하는 경우들이 있다. Long read 분석 기술이나, 생물정보 분석기술이 더 발전해야 하고, 또 한국인이 더 많이 포함된 변이 데이터베이스가 필요하다는 의미이기도 하다.
2. WGS를 하더라도 Human Phenotype Ontology(HPO)를 이용한 분석이 필요하다. HPO기반 분석이나 software들이 많이 개발되고 있으므로 HPO를 이용하는 것이 실질적인 표준 분석방법으로 사용될 것으로 예상된다.
3. 단일 환자보다 환자의 부모를 포함한 trio 분석이 더욱 더 높은 발견율을 보인다. 실제 1차년도의 경험으로는 단일 환자보다 trio 분석 발견율이 두 배 높다. 특히 발달지체 질환 등에 WGS을 해야 될 경우, 가능하면 trio 분석이 필요하다.
4. Coding sequence 이외 noncoding sequence에 대한 분석은 점점 더 일상화되고 필요해질 것으로 생각한다. 여기에는 deep intron pathogenic variant나 structural variation 등이 해당된다. 복합질환의 분석을 같이 해야 한다면, 특히 전술한 바와 같이 noncoding sequence 분석이 기본이다.
5. 따라서, 우리나라는 임상 검사로서 whole exome sequencing (WES)을 건너뛰고, WGS으로 바로 이행하지 않을까 전망한다. Panel based NGS 후 시행한 WES에는 추가로 얻는 진단정보가 그렇게 많지 않다. WGS는 noncoding sequence 정보나 structural variation, mitochondria 염기서열정보를 얻을 수 있다. WES와 WGS 중에서 임상 진단을 위해서는 WGS가 압도적으로 선호가 될 것으로 보인다.
6. 생물정보 분석에 대한 체계나 지식은 점점 더 많이 필요해질 것으로 보인다. 평균 1명의 BAM file x30 coverage 기준으로 100GB의 대량의 데이터가 발생하며, 한국인의 1인당 평균 변이 수가 약 350만개 정도임을 감안하면 이들 data를 다루고 병적 변이를 판독하는 일은 전산체계 구축과 생물정보 인력의 투자를 필요로 한다. 효과적인 WGS검사의 수행과 판독을 위해서 기존의 유전검사 판독시스템에 이들 시스템과 투자가 인정되는 의료 수가나 제도적 보완이 필요하다.
맺는 말 한국의 바이오 빅데이터 사업은 시범 사업을 시작한 지 만 2년도 되지 않은 상태이다. 그럼에도 WGS가 희귀 질환의 유전 진단에 중요한 역할을 한다는 것을 실제 경험으로 알게 해주고 있으며, 나아가 희귀질환과 복합질환을 막론하고 일상적인 임상 검사 수단으로서 WGS가 필요하다는 것을 보여준다.
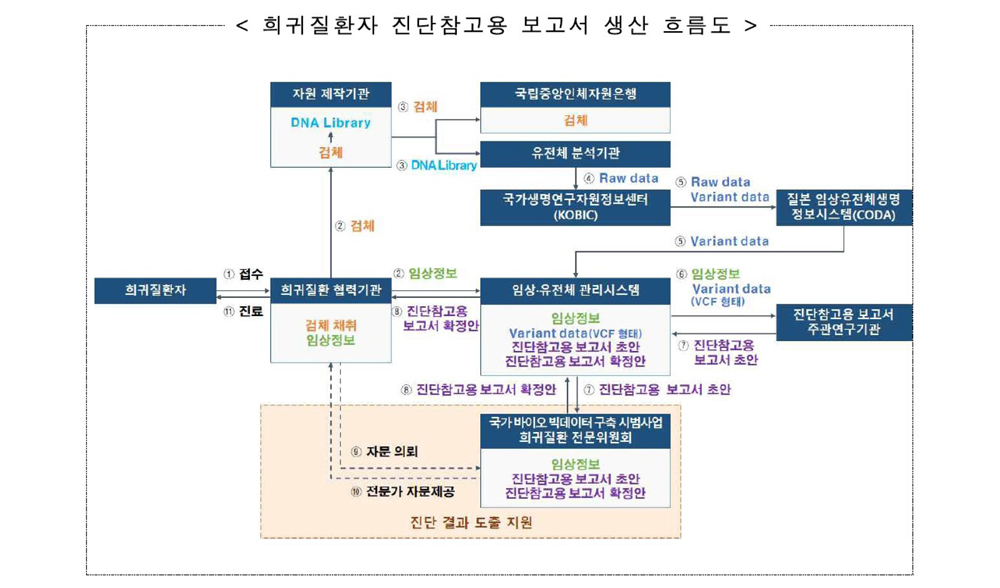 Fig 1. 국가 바이오 빅데이터 사업 희귀질환 환자 과제 흐름도
Fig 1. 국가 바이오 빅데이터 사업 희귀질환 환자 과제 흐름도
[참고 문헌 및 website]
1. Claussnitzer M, Cho JH, Collins R, Cox NJ, Dermitzakis ET, Hurles ME, Kathiresan S, Kenny EE, Lindgren CM, MacArthur DG, North KN, Plon SE, Rehm HL, Risch N, Rotimi CN, Shendure J, Soranzo N, McCarthy MI. A brief history of human disease genetics. Nature. 2020 Jan;577(7789):179-189.
2. All of Us research program. https://allofus.nih.gov/
3. Genomics England. https://www.genomicsengland.co.uk/
4. 국가통합 바이오빅데이터 구축사업. https://bighug.kdca.go.kr/bigdata/

