연세의대
1. 서론
DNA와 RNA 시퀀싱은 현대 생물학 연구에 필수적인 도구로 자리잡았으며, 지난 20년간 차세대 염기서열 시퀀싱 (next-generation sequencing; NGS) 플랫폼은 다양한 유기체의 게놈과 전사체 해독에 중추적인 역할을 해왔다. 주요 제조업체인 Illumina의 플랫폼과 비교하여 유사한 용량, 다용도성, 정확도를 제공할 수 있는 대안적 플랫폼의 부재가 있었지만, 2022년에는 Element Biosciences, Singular Genomics, Ultima Genomics, MGI를 포함한 여러 새로운 NGS 플랫폼 제조업체들이 시장에 진입하면서 다양한 선택지가 제공되고 있다. 이중 Element Biosciences의 AVITI 시스템에 관한 정보와 사용 경험을 나누려고 한다.
2. 본론
AVITI 시스템의 기술적 특징
AVITI System은 Avidity Sequencing™이라고 알려진 새로운 기술을 사용한다. 선형 라이브러리를 splint oligo와 결합하여 원형화하고 플로우 셀에서 rolling circle amplification을 통해 증폭하여 중합체 템플릿 가닥을 형성한다(그림1-가). 각 가닥은 원래 라이브러리의 수백 개 복사본을 포함하는 polony를 형성하며 이는 높은 신호 대 잡음비를 가진 시퀀싱을 가능하게 한다. Sequencing-by-synthesis 방식을 활용하는 Illumina 플랫폼들과 비교하여 PCR-free 전체 유전체 시퀀싱에서 중복 시퀀싱 리드 (duplicate sequence) 생성이 적은 것을 검증한 연구들이 보고된 바 있으며, 이론상 인덱스 호핑을 최소화 시킬 수 있다. 또한 어려운 호모폴리머 및 반복 영역에서 소프트 클립된 리드가 적게 생성되는 것을 확인할 수 있다.
 그림1
그림1
(가) Rendering of a single avidite bound to a DNA polony via polymerase-mediated selection [figure from [1] Arslan S, et al. (2024). Nat Biotechnol 42(1):132-138] (나) AVITI System platform specifications (다) AVITI System workflows
플로우 셀과 처리량
AVITI 시스템은 독립적으로 작동할 수 있는 두 개의 플로우 셀을 동시에 사용할 수 있으며, 각 플로우 셀은 2×150bp 기준으로 10억 개의 염기가닥을 읽을 수 있다 (그림1-나). 또한, 2 x 75bp, 2 x 150bp, 2 x 300bp 길이의 염기서열을 읽을 수 있으며, 사용자의 필요에 따라 high, medium, low 용량의 키트를 제공한다. 높은 정확도를 달성하기 위해 시퀀싱 시간이 다소 길어질 수 있으므로, 실험을 계획할 때 이 점을 유의해야 한다.
라이브러리 제작 워크플로우
AVITI 시스템의 라이브러리 제작에는 “Elevate”와 “Adept” 두 가지 워크플로우가 있다(그림1-다). 이 두 워크플로우는 라이브러리 제작의 효율성과 유연성을 높이기 위해 설계되었다. Elevate는 AVITI 시스템의 고유 방식으로 고품질의 라이브러리 제작을 목표로 한다. 그에 반해 Adept는 타회사 라이브러리를 PCR-plus 또는 PCR-free로 변환하여 분석하는 방식이다. PCR 단계가 포함된 Adept의 경우 여러 패널을 동시에 실험할 때 genome 크기에 따라 output 크기가 변경될 수 있어 최종 산물의 DNA양을 정량적으로 확인하는 것이 추천된다.
데이터 품질
Base2Fastq라는 프로그램을 사용하여 FASTQ 파일을 생성할 수 있으며, 한 플로우 당 최대 300Gb까지 분석 가능하다. FASTQ 형식은 Illumina와 비슷하게 R1, R2로 분류되며 Illumina 파이프라인에 동일하게 적용해도 된다. Adept로 Illumina 라이브러리를 변환하였을 때 Gb당 읽힌 염기서열 수가 Illumina에 비해 AVITI에서 2/3정도 낮은 것을 확인했다 (그림2-가). 이는 라이브러리 변환 시 추가되는 시퀀스에 대한 영향 또는 FASTQ 압축 방식 차이일 수 있다. AVITI 시스템은 높은 데이터 품질을 제공하며, Illumina보다 duplicate read sequence 비율이 평균 6.47% 낮고, 데이터 품질은 >Q30가 96.2%, PCR-free 라이브러리의 경우 >Q40가 85.4%라는 결과를 보였다 (그림2-나). NA12878 표준물질을 이용한 실험에서 sensitivity는 99.6%, specificity는 99.9%로 나타났으며, heterozygous SNP의 variant allele frequency의 평균은 AVITI에서 51.09% (SD 6.05%), NovaSeq에서 51.28% (SD 5.01%)가 관찰됐다.
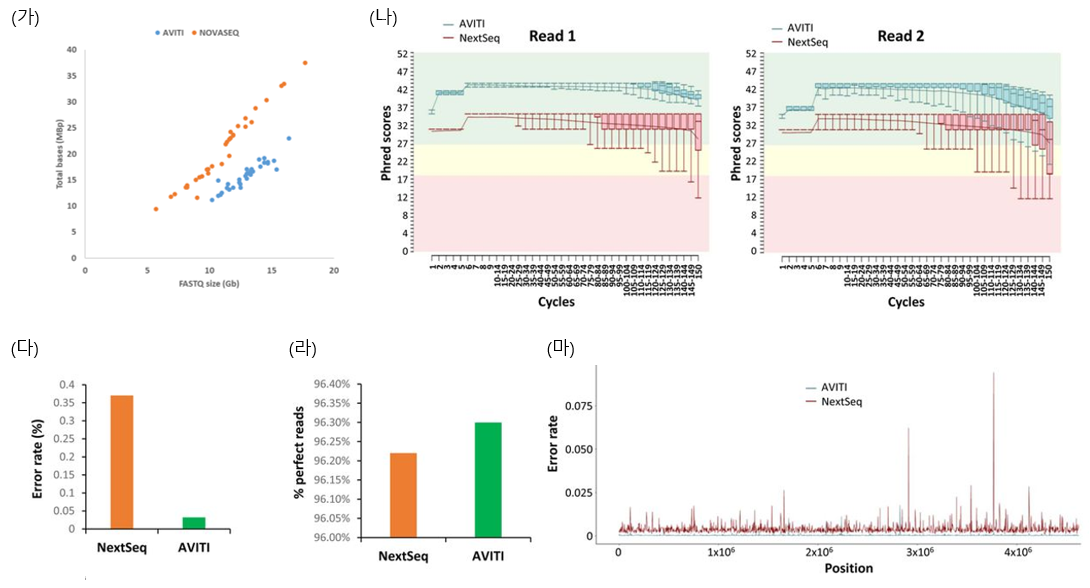 그림2
그림2
(가) Bases sequenced per FASTQ size (나) Phred score distribution cycle by cycle of AVITI and NextSeq550 (다) Percent error rate of the mapped reads from AVITI and NextSeq550. (라) Percentage of the mapped reads from AVITI and NextSeq550. (마) Histogram of 2000 loci error rate in the E. coli genome by AVITI and NextSeq550. [figures from [4] Liu S, et al. (2024) bioRxiv 590136]
비용 효율성
AVITI 시스템은 Illumina의 NovaSeq 및 NextSeq와 비교했을 때 초기 투자 비용, 운영 비용 등 여러 면에서 경제적인 장점을 제공한다. 미국시장에서 AVITI 장비의 가격은 NextSeq의 약 86% 수준으로 설정되어 있으며, 기가베이스(Gb)당 분석 비용 역시 NextSeq에 비해 50-70% 가량 저렴하다. 또한, NovaSeq SP와 비교했을 때도 30% 저렴하다고 알려져, 한국으로 유통되는 과정에서 가격 변동은 가능하나, 비용 효율성 면에서 AVITI 시스템은 여러 연구실에 매력적인 선택지가 될 수 있다.
미래 전망
NGS 장비 분야의 후발주자인 AVITI는 사용자 편의성을 중심으로 지속적으로 새로운 분석 방법을 개발해왔으며, 2024년에 선보일 다양한 기술들을 준비하고 있다. Cloudbreak Freestyle은 타사 라이브러리를 AVITI 장비 내에서 직접 변환 및 분석할 수 있게 함으로써 사용자의 실제 작업 시간을 단축시킬 것으로 기대된다. UltraQ는 AVITI 시스템의 고정밀 시퀀싱 솔루션으로, 낮은 오류율과 높은 정확도가 필수적인 응용 분야에 최적화되어 있으며, Q50를 목표로 연구가 진행 중이다. Trinity는 바코딩된 라이브러리에 대해 probe hybridization, enrichment, sequencing 등의 과정을 장비 내부에서 자동으로 처리하는 기술이다. 또한, AVITI는 1,500bp 길이의 긴 염기서열 처리가 가능하여, 구조적 변이나 융합 유전자 검출에 있어 우위를 점할 수 있다. AVITI24는 DNA, RNA, 단백질, 형태학을 24시간 내에 동시에 분석할 수 있는 멀티오믹스 분석 방법으로, AVITI 시스템의 성능과 유연성을 크게 강화시키는 새로운 기능들을 통해 다양한 연구 요구사항을 충족시킬 것으로 기대된다.
Element Biosciences의 AVITI 시스템은 Avidity Sequencing 기술을 통해 높은 데이터 품질과 경제성을 제공하며, 다양한 연구 요구사항을 충족시키기 위해 지속적으로 발전하고 있다. 이러한 새로운 기능들은 연구자들에게 더 많은 선택지와 효율성을 제공하여 현대 생물학 연구에 큰 기여를 할 것으로 기대된다.
[References]
[1] Arslan S, Garcia FJ, Guo M, Kellinger MW, et al. Sequencing by avidity enables high accuracy with low reagent consumption. Nat Biotechnol. 2024 Jan;42(1):132-138.
[2] Caroll A, Kolesnikov A, Cook DE, Brambrink L, et al. Accurate human genome analysis with Element Avidity sequencing. bioRxiv. 202308.11.553043
[3] Li JH, Findley K, Pickrell JK, Blease K, Zhao J, Kruglyak S. Low-pass sequencing plus imputation using avidity sequencing displays comparable imputation accuracy to sequencing by synthesis while reducing duplicates. G3 (Bethesda). 2024 Feb 7;14(2):jkad276.
[4] Liu S, Obert C, Yu YP, et al. Utility Analyses of AVITI Sequencing Chemistry. bioRxiv. 2024.04.18.590136.
[5] Majidian S, Agustinho DP, Chin CS, Sedlazeck FJ, Mahmoud M. Genomic variant benchmark: if you cannot measure it, you cannot improve it. Genome Biol. 2023 Oct 5;24(1):221.

