Nortable Research
An International, Multicentered, Evidence-Based Reappraisal of Genes Reported to Cause Congenital Long QT Syndrome
사용자 경험 최신 보험정보분당서울대병원
미국 National Institutes of Health (NIH)의 지원으로 설립된 The Clinical Genome Resource (ClinGen)은 특정 유전자와 질병의 연관성을 평가하고 이를 기반으로 환자, 임상의사 및 연구자들에게 실질적으로 도움이 되는 정보를 생성하는 기관입니다. ClinGen은 유전자와 그 변이의 임상적 관련성을 정의할 수 있도록 표준 프로세스를 개발하여 최근 연구 영역에서 진료 영역으로 넘어온 NGS가 활성화된 임상 유전자 검사 결과의 해석에 도움을 주고 있습니다.
최근 이 ClinGen working group에서 정리한 Long QT Syndrome (LQTS)과 관련된 유전자들의 유전자-질병의 타당성에 대한 검증 결과가 Circulation에 실렸습니다.1 LQTS는 대표적인 유전성 부정맥 질환으로, 1995년 처음으로 KCNH2, SCN5A 유전자가 이를 일으키는 원인이 된다는 것이 밝혀진 후 지난 25년간 17개 가량의 유전자들이 이 질환의 원인 유전자로 보고되었습니다. LQTS의 유병률은 1:2000 정도로 알려져 있으며, 유전자 검사를 시행해 변이가 확인될 경우 치료제의 선택에 도움을 받을 수 있고 아직 증상이 발현되지 않은 가족에서의 검사도 가능하여 sudden cardiac death의 위험을 현저히 감소시킬 수 있다는 장점이 있기에 유전자 검사를 시행하는 것이 임상적으로 큰 도움이 되는 질환입니다. 그러나 동시에 잘못된 유전자 검사 결과는 불필요한 불안감, 라이프스타일 변화, 보험 혹은 직업 관련 이슈, 나아가 잘못된 치료로도 연결될 수 있다는 점에서 주의를 요하고 있습니다. 이에 ClinGen에서는 임상의사, 유전학자, 유전상담가, 연구자들이 모여 International, Multicentered LQTS ClinGen Working Group을 조직해 현재까지 발표된 LQTS의 원인 유전자들을 대상으로 표준화된 프레임워크2를 이용하여 유전자-질병 타탕성을 재평가했습니다.
대상이 된 유전자는 PubMed에서 검색된 17개의 LQTS 관련 유전자입니다. (AKAP9, ANK2, CALM1, CALM2, CALM3, CAV3, KCNE1, KCNE2, KCNH2, KCNJ2, KCNJ5, KCNQ1, SCN4B, SCN5A, SNTA1, TRDN). 이 중 전형적 LQTS의 대표적인 원인 유전자로 알려져 있던 KCNQ1, KCNH2, SCN5A는 질병과의 연관성에서 확실한 증거를 보여 “Definitive”로 분류되었고, CALM1, CALM2, CALM3, TRDN의 경우에는 비전형적 LQTS에 대하여 “Definitive” 및 “Strong”으로 분류되었습니다. CACNA1C는 “Moderate”로 분류되었으며 나머지 9개의 유전자는 근거 부족으로 평가되어 “Disputed” 혹은 “Limited”로 분류되었습니다 (Table 1). 이 중에서 KCNE1과 KCNE2의 경우, 약물 등의 2차적인 원인이 있는 경우 발생하는 후천성 LQTS의 predisposing risk alleles로 분류될 수 있는 강한 근거가 있는 것으로 평가되어 이에 대해서는 “Strong”으로 분류되었습니다. 또한 CACNA1C (Timothy Syndrome)과 KCNJ2 (Andersen-Tawil Syndrome)의 경우에는 prolonged QT interval과 ventricular arrhythmia가 동반되는 각각의 증후군에 한하여 “Definitive” 한 것으로 결론을 내렸습니다.
“Disputed” 혹은 “Limited”로 분류된 유전자들을 자세히 살펴보면 대부분 근거가 부족했습니다. 해당 유전자들은 주로 후보유전자 접근방법(candidate gene approach)이 적용된 연구에서 추정된 경우였거나, 검출된 염기변이가 추후 일반인구집단에서 예상보다 높은 비율로 검출된 경우, 가계 내 유전자 분리(segregation) 정보가 없는 경우, 혹은 특정 염기변이의 검출 비율이 대조군과 비교해 통계적으로 유의한 차이를 보이지 않는 경우 등이었습니다. “Disputed”로 분류된 6개의 유전자 (AKAP9, ANK2, KCNE2, KCNJ5, SCN4B, SNTA1) 중 4개(AKAP9, KCNE2, SCN4B, SNTA1)에 대한 논문들은 논문들은 후보유전자 접근방법(candidate gene approach)으로 원인 유전자를 추정해 나갔으며, 검출된 변이에 대해 유의한 segregation을 보이지는 못했습니다.
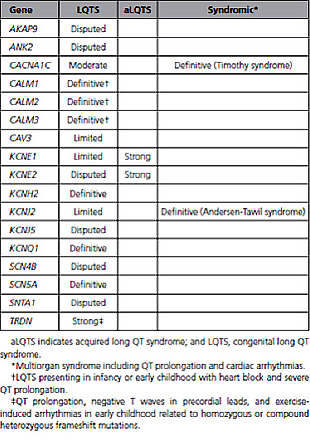
Table 1. Classification of genetic evidence for genes previously reported as causing LQTS
ANK2와 KCNJ5 유전자의 경우에는 대가족을 대상으로 한 연관분석(linkage analysis)를 시행한 초기 논문들을 근거로 하였으나, 대상 부위가 16 Mbp 정도로 넓은 범위이며 그 안에 포함된 다른 유전자들에 대한 평가가 되지 않았습니다. 또 해당 유전자에서 검출된 염기변이들 또한 일반인구집단에서 상당히 높은 비율로 검출되는 것이 추후 확인되어 그 의미를 논하기 어려웠습니다. “Limited”로 분류된 3개의 유전자(CAV3, KCNE1, and KCNJ2) 역시 후보유전자 접근방법(candidate gene approach)을 기반으로한 문헌을 근거로 하고 있으나 이에 대한 추가 근거는 제한적이었습니다. CAV3의 경우에는 검출된 염기변이가 de novo로 보고되었으나 부모의 임상상 및 혈연관계 확인 등은 언급되어 있지 않았습니다. KCNE1의 경우, 후천성 LQTS과의 연관성을 보인다는 이유에서 2차 자극이 없는 상태에서도 LQTS를 일으킬 가능성이 있다고 보고되었으나 이에 대한 추가 연구는 없었습니다. 단, KCNE1의 homozygous 혹은 compound heterozygous 변이들이 Jervell and Lange-Nielson syndrome에서 검출된 것으로 보고된 케이스들이 있어, 해당 유전자의 변이들이 상염색체열성 패턴의 유전 형태를 가질 수 있음은 고려되어야 할 것 같습니다.

“Definitive” 혹은 “Strong”으로 분류된 유전자들 가운데에도 임상상으로는 구분되는 점이 있었습니다. 오래전부터 LQTS의 원인 유전자로 알려져 있던 KCNQ1, KCNH2, SCN5A, 이 세 유전자의 경우에는 전형적인 LQTS의 임상상이 주로 확인되었던 것에 비해, calmodulin 관련 유전자(CALM1, CALM2, CALM3)의 경우에는 비전형적 LQTS 양상을 보이는 것으로 확인되었습니다. TRDN 유전자 역시 비전형적 LQTS의 특징을 갖는 것으로 분류되었습니다.
여기서 언급되는 비전형적인 LQTS의 특징들로는 상염색체 열성으로 유전되는 유전 양식과 3살 이하의 어린 나이에 발병한다는 특징 외에도 precordial leads의 negative T wave를 보인다는 ECG 소견들이 알려져 있습니다. 정리하자면, 기존에는 LQTS의 유전적 지형도(genetic landscape) 를 설명할 때, 대표적인 유전자 3개가 환자들의 75%-95%에서 검출되고 나머지 마이너 유전자들이 드물게 검출되는 것으로 설명되었으나 이것을 수정할 필요가 생긴 것입니다. 즉, 기존의 대표적인 유전자 3개(KCNQ1, KCNH2, SCN5A)는 전형적 LQTS의 원인 유전자이며, CALM1, CALM2, CALM3 및 TRDN은 비전형적인 LQTS의 원인 유전자로 분류하는 것이 더 적절해 보입니다.
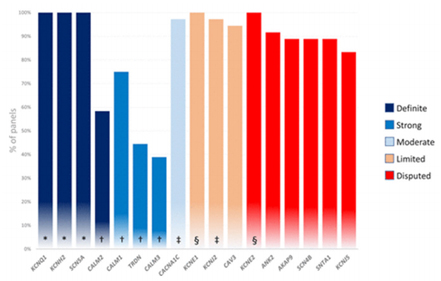 Figure 1. Composition of LQTS-specific genetic panels
Figure 1. Composition of LQTS-specific genetic panels
이런 결과를 바탕으로 ClinGen working group 에서는 현재 여러 임상검사실에서 시행되고 있는 LQTS 관련 유전자 패널들을 조사하였습니다. 총 36개의 LQTS 관련 유전자 패널들을 조사한 결과, 5개의 유전자(KCNQ1, KCNH2, SCN5A, KCNE1, KCNE2)는 조사된 모든 패널에 포함되어 있었고, “Disputed/Limited”로 분류된 유전자들을 포함하고 있는 경우가 83%-100%, 비전형적인 LQTS의 “Definitive/Strong” 유전자인 CALM1, CALM2, CALM3, TRDN을 포함하는 경우는 39%-75% 밖에 되지 않았습니다 (Figure 1). 연구적인 차원에서는 질병과의 연관성이 명확하지 않은 유전자들에 대해 군집 분석(segregation analysis) 데이터 등을 추가해 유전자-질병 타탕성을 밝히는 것이 매우 중요합니다. 그러나 임상적으로 활용될 유전자패널 검사에서는 이런 유전자들에 대한 검사를 시행할 경우, 해당 유전자에서 검출되는 염기변이들에 대한 해석이 어려워 결국 많은 수의 염기변이들이 variants of uncertain significance (VUS)로 보고될 가능성이 높습니다. 이는 검사를 의뢰한 의사들과 환자들에게 불확실성을 전하며, 동시에 불필요한 비용과 불안감을 증가시킬 가능성 이 높아 해당 유전자들을 임상적인 유전자패널 검사에 포함시키는 것에 대해서는 주의가 필요합니다. 정밀 의료의 임상적인 적용을 위해 유전자-질병 타탕성을 주기적으로 검토하는 것이 중요할 것으로 생각됩니다.
1. Adler A, Novelli V, Amin AS, et al. An International, Multicentered, Evidence-Based Reappraisal of Genes Reported to Cause Congenital Long QT Syndrome. Circulation 2020;141:418-428.
2. Strande NT, Riggs ER, Buchanan AH, et al. Evaluating the Clinical Validity of Gene-Disease Associations: An EvidenceBased Framework Developed by the Clinical Genome Resource. Am J Hum Genet 2017;100:895-906.

